Определение клубочков на больших изображениях ткани почек человека средствами сегментации [распознавание]
Ссылка на предыдущую статью с обучением модели
Основная проблема при распознавании больших изображений - это чтение их из файла. Ссылки на ноутбуки, где представлено распознавание вместе с submit, можно найти в предыдущей статье, которую нужно прочитать, прежде, чем переходить к этой.
Инициализация
Загрузка параметров из результатов обучения:
mod_path = '/kaggle/input/hubmap-efficientunet-512-2048-test/' # путь до моделей
import yaml
import pprint
with open(mod_path+'params.yaml') as file:
P = yaml.load(file, Loader=yaml.FullLoader)
pprint.pprint(P)
WINDOW = 2048 # окно просмотра
MIN_OVERLAP = 1024 #пересечение окон
NEW_SIZE = P['DIM'] #размер, к которому приводится окно просмотра
Результат у меня был такой:
{'BACKBONE': 'efficientnetb5',
'BATCH_COE': 8,
'DIM': 512,
'DISPLAY_PLOT': True,
'EPOCHS': 60,
'LR': 0.00025,
'NFOLDS': 4,
'SEED': 0,
'VERBOSE': 1}
Импорт библиотек:
! pip install ../input/kerasapplications/keras-team-keras-applications-3b180cb -f ./ --no-index -q
! pip install ../input/efficientnet/efficientnet-1.1.0/ -f ./ --no-index -q
import numpy as np
import pandas as pd
import os
import glob
import gc
import json
import numba
import rasterio
from rasterio.windows import Window
import pathlib
from tqdm.notebook import tqdm
import cv2
import tensorflow as tf
import efficientnet as efn
import efficientnet.tfkeras
Загрузка моделей
Загрузка моделей очень проста:
fold_models = [
for fold_model_path in glob.glob(mod_path+'*.h5'):
fold_models.append(tf.keras.models.load_model(fold_model_path,compile = False))
Основной цикл работы с большими изображениями
В цикле происходит перебор и чтение tiff файлов, которые нужно распознать, после чего внутренний цикл "пробегается" окном по всему изображению, распознавая маску с клубочками. Перекодирование в формат понятный kaggle не приведено. Часть функций, которые вызываются в цикле будет приведена ниже.
p = pathlib.Path('../input/hubmap-kidney-segmentation')
for i, filename in tqdm(enumerate(p.glob('test/*.tiff')),
total = len(list(p.glob('test/*.tiff')))):
print(f'{i+1} Predicting {filename.stem}')
dataset = rasterio.open(filename.as_posix(), transform = identity)
print(dataset.dtypes)
slices = make_grid(dataset.shape, window=WINDOW, min_overlap=MIN_OVERLAP)
preds = np.zeros(dataset.shape, dtype=np.uint8)
if dataset.count != 3:
print('Image file with subdatasets as channels')
layers = [rasterio.open(subd) for subd in dataset.subdatasets]
th = 0.4
pbar = tqdm(total=len(slices))
for (x1,x2,y1,y2) in slices:
if dataset.count == 3:
image = dataset.read([1,2,3],
window=Window.from_slices((x1,x2),(y1,y2)))
image = np.moveaxis(image, 0, -1)
else:
image = np.zeros((WINDOW, WINDOW, 3), dtype=np.uint8)
for fl in range(3):
image[:,:,fl] = layers[fl].read(window=Window.from_slices((x1,x2),(y1,y2)))
image = cv2.resize(image, (NEW_SIZE, NEW_SIZE),interpolation = cv2.INTER_AREA)
image = cv2.cvtColor(image, cv2.COLOR_RGB2BGR)
image = np.expand_dims(image, 0)
pred = None
for fold_model in fold_models:
if pred is None:
pred = np.squeeze(fold_model.predict(image))
else:
pred += np.squeeze(fold_model.predict(image))
pred = pred/len(fold_models)
pred = cv2.resize(pred, (WINDOW, WINDOW))
preds[x1:x2,y1:y2] |= mask_filter2((pred > th).astype(np.uint8))
pbar.update(1)
print(np.sum(preds))
del preds
gc.collect();
Чтение заголовка tiff файла производится с помощью библиотеки rasterio. С помощью функции make_grid изображение разбивается на условные кусочки (slices). А для всего изображения создается матрица preds, в которую будут записаны результаты распознавания. Поскольку tiff файлы могут быть не просто цветными (RGB), а трёхслойными (где каждый канал изображения в отдельном слое), то по условию if dataset.count != 3 читаются слои.
Далее идёт цикл перебора кусочков for (x1,x2,y1,y2) in slices, в котором читаются данные для кусочка в изображение формата numpy. Которое затем функциями OpenCv приводятся к нужному размеру и BGR формату. После чего изображение распознается 4-мя моделями (fold_model.predict(image)), результат при этом суммируется, а после нормализуется делением на количество моделей. Затем происходит растяжение к исходному размеру и записывается в матрицу preds, используя функцию mask_filter2:
def mask_filter2(img):
w = int(img.shape[0]*0.2)
h = int(img.shape[1]*0.2)
img[0:h, 0:img.shape[0]] = 0
img[img.shape[1]-h:img.shape[1], 0:img.shape[0]] = 0
img[h:img.shape[1]-h,0:w]=0
img[h:img.shape[1]-h,img.shape[0]-w:img.shape[0]]=0
return img
Назначение этой функции - обрезать края изображения, поскольку на них могут быть ошибки. А так как мы распознаем с перекрытием, то эти удаленные участки будут распознаны в других частях.
Функция make_grid имеет следующий вид:
def make_grid(shape, window=256, min_overlap=32):
"""
Return Array of size (N,4), where N - number of tiles,
2nd axis represente slices: x1,x2,y1,y2
"""
x, y = shape
nx = x // (window - min_overlap) - 1
x1 = np.linspace(0, x, num=nx, endpoint=False, dtype=np.int64)
x1[-1] = x - window
x2 = (x1 + window).clip(0, x)
ny = y // (window - min_overlap) - 1
y1 = np.linspace(0, y, num=ny, endpoint=False, dtype=np.int64)
y1[-1] = y - window
y2 = (y1 + window).clip(0, y)
slices = np.zeros((nx,ny, 4), dtype=np.int64)
for i in range(nx):
for j in range(ny):
slices[i,j] = x1[i], x2[i], y1[j], y2[j]
return slices.reshape(nx*ny,4)
Заключение
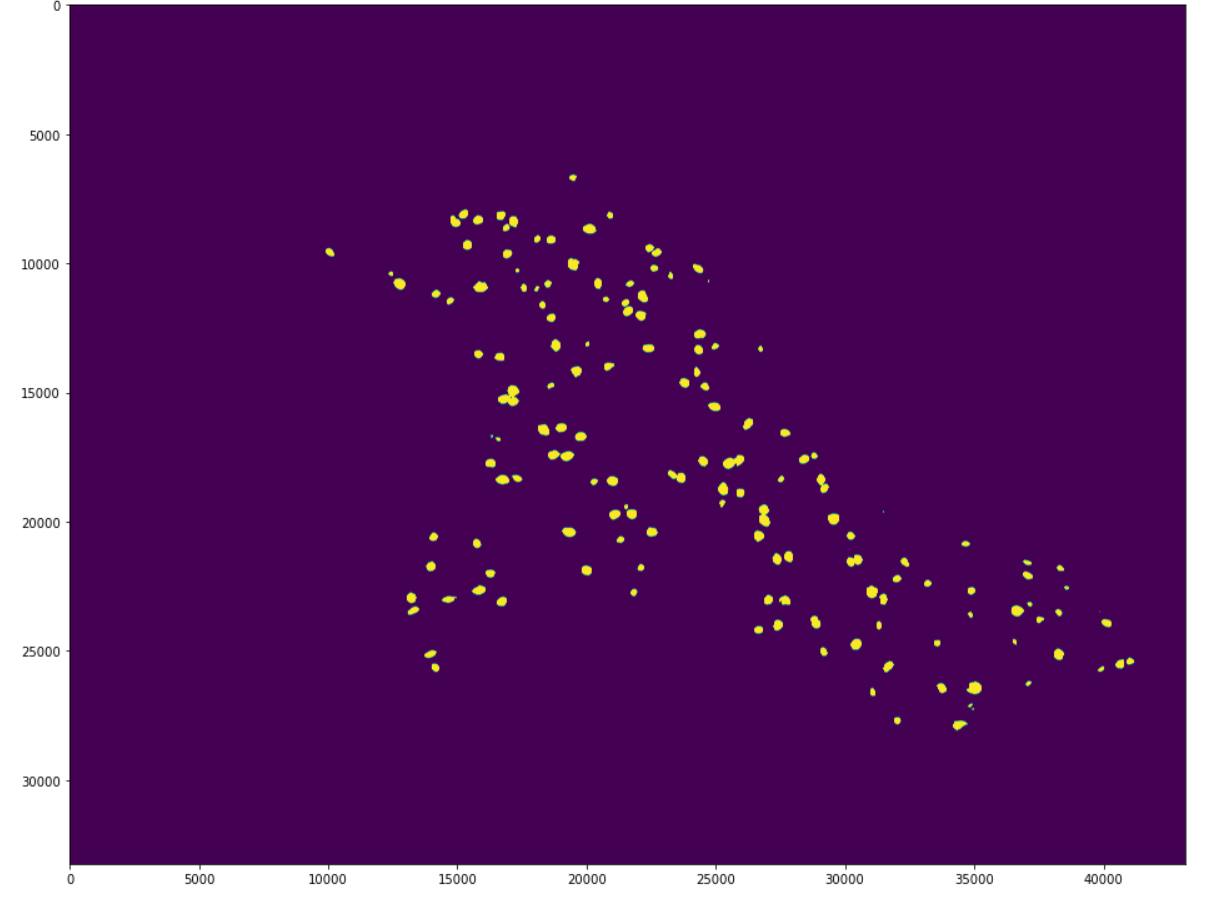
Если вы хотите посмотреть полученное предсказание целиком, то можно вывести preds для одного изображения:
import matplotlib.pyplot as plt
from PIL import Image
plt.figure(figsize=(16, 16))
plt.imshow(Image.fromarray(preds))
plt.show()
Результат: